Bonjour à tous,
Aujourd’hui, je vais vous parler de formattable.
This package is designed for applying formatting on vectors and data frames to make data presentation easier, richer, more flexible and hopefully convey more information.
Nous allons voir comment utiliser cette librairie pour interpréter nos données en un coup d’oeil, à l’aide de quelques lignes de code (vous pouvez suivre les indications ci-dessous ou aller regarder tout le code disponible sur git). Avant d’aller plus loin, j’aimerais préciser que cette librairie est généralement utilisée pour afficher des tableaux dans un document dynamique (comme un rapport) généré par une autre librairie, comme R markdown. Mais ici, j’ai choisi de restreindre le sujet de cet article à l’utilisation de la première librairie seulement. La seconde fera peut-être l’objet d’un second article. En attendant, vous pouvez trouver de l’aide sur la page web de R markdown ou sur le web (comme sur stackoverflow).
Données de travail:
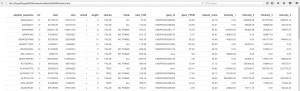
Je vous propose de travailler sur des données publiées par Céline M. Laumont and et al. dans l’article Global proteogenomic analysis of human MHC class I-associated peptides derived from non-canonical reading frames, avec le tableau trouvé dans « Supplementary Data 2 ». Ce tableau contient des données sur des peptides cryptiques au niveau génomique et proéomique.

Figure 1. Une version Excel des premières lignes du tableau.
Créer votre premier tableau formattable :
Tout d’abord, il faut commencer par:
- installer la librairie dans votre R (ou Rstudio):
- charger la librairie et les données:
install.packages("formattable") # Charger la librarie avant de l'utiliser
library(formattable)
# Charger les données
tab = read.table("./Supplementary_table_2_CrypticMAPs.txt", header=T, sep='\t', stringsAsFactors=F)
# Sélectionner quelques colonnes comme dans le tableau Excel
tab_reduce = tab[, c(1, 2, 3, 4, 6, 5, 7, 9, 11, 18, 15, 19, 22, 23, 24, 25)]
# Changer le nom de certaines colonnes pour diminuer leur taille
colnames(tab_reduce) = c("peptide_sequence", "chr", "start", "stop", "strand",
"length", "spliced", "frame", "best_IC50", "gene_id", "gene_FPKM",
"mascot_score", "intensity_1", "intensity_2", "intensity_3", "intensity_4"
)
Nous pouvons maintenant créer notre premier tableau formattable en appelant directement la fonction formattable:
# Créer un tableau formattable
widget_formattable = formattable(tab_reduce)
# Afficher dans un navigateur web ou dans RStudio (si vous l'utilisez)
widget_formattable
Figure 2. Résultat visuel après l’exécution de formattable. Toutes les données sont disponibles en les faisant défiler dans la page web!
Corriger le format de la colonne ‘gene_FPKM’:
Dans la librairie formattable, il existe plusieurs fonctions pour formater les vecteurs numériques, comme: percent, comma, currency, accounting and scientific. Ces fonctions créent des vecteurs numériques formatés à l’aide de quelques paramètres et de règles pré-définies. Pour plus de détails, je vous invite à regarder cette page.
Dans notre cas, la colonne ‘gene_FPKM’ est automatiquement chargée comme un vecteur de ‘chr’ par read.table à cause des quelques valeurs manquantes (‘none’). Il est nécessaire de modifier la colonne en un vecteur numérique avant d’aller plus loin.
# Un message d'erreur nous avertit qu'il y a des valeurs 'None' et qu'elles seront remplacées par des NA. -> C'est correct.
tab_reduce$gene_FPKM = as.numeric(tab_reduce$gene_FPKM)
# Utiliser 'accounting' pour formater les valeurs numériques
tab_reduce$gene_FPKM = accounting(tab_reduce$gene_FPKM)
Afficher des élément graphiques en fonction des données de chaque colonne:
Commençons par utiliser les fonctions déjà présentes dans la librairie, color_tile(), color_bar(), area() and normalize_bar(), pour mettre en valeur les données et ainsi faciliter la comparaison de leur magnitude (voir Figure 3).

Figure 3. Ajout d’éléments graphiques pour faciliter la comparaison des différentes valeurs.
Dans la Figure 3, il faut noter que nous souhaitons mettre en valeur les faibles scores de la colonne IC50, contrairement aux autres colonnes: gene FPKM, mascot_score and intensity. C’est aussi important de savoir que les intensités doivent être comparées par peptide. En conséquence, l’élément graphique doit partager la même échelle pour toutes les colonnes d’intensité, cette fonctionnalité sera réalisée par la fonction area.
Toute ces contraintes peuvent être appliquées en quelques lignes en ajoutant une liste d’éléments graphiques identifiés par le même nom que la colonne où l’on veut les afficher: ‘best_IC50’, ‘gene_FPKM’, ‘mascot_score’ et les quatre ‘intensity_x‘.
# Utilisation de fonctions 'built-in' pour afficher des élements graphiques.
widget_formattable = formattable(tab_reduce, list(
best_IC50 = color_tile('lightblue', 'white'),
# Nous avons besoin de mettre na.rm=TRUE pour gérer les valeurs NA présentes dans les données
gene_FPKM = color_bar('red', na.rm = TRUE),
mascot_score = color_tile('white', 'orange'),
area(col=c(intensity_1, intensity_2, intensity_3, intensity_4)) ~ normalize_bar("pink")
))
widget_formattable
Pour aller plus loin:
La librairie formattable permet d’ajouter des éléments graphiques en ajoutant des éléments HTML, principalement à l’aide des balises <span>, en utilisant une fonction générique ‘formatter‘. Un exemple simple de l’utilisation de cette fonction peut être fait pour afficher en gras le sens du peptide (strand) en ajoutant dans la liste:
strand = formatter('span', style=style(font.weight = "bold"))Il est aussi possible d’en faire une utilisation plus complexe en utilisant, ifelse pour créer vos propres fonctions:
- pour changer le visuel d’une colonne en fonction d’une autre colonne:
none_formatter <- function() {
formatter("span",
style = ~ style(color = ifelse(gene_id == "none", "blue", "black"))
)
}
- pour afficher des icônes de différentes couleurs représentant des valeurs booléennes:
icon_formatter <- function() {
formatter("span",
style = x ~ style(color = ifelse(x, "green", "red")), x ~ icontext(ifelse(x, "ok", "remove"), "")
)
}
- modifier les fonctions existantes pour supprimer un effet de bord sur les valeurs 'NA' (voir Figure 3):
color_bar_NA <- function(color = "lightgray", fun = "proportion", ...) {
fun <- match.fun(fun)
replace_na <- function(fun, x, ...) {
x[which(is.na(x))] = 0
return(fun(as.numeric(x), ...))
}
formatter("span",
style = function(x) style(
display = "inline-block",
direction = "rtl",
"border-radius" = "4px",
"padding-right" = "2px",
"background-color" = csscolor(color),
width = percent(replace_na(fun, x, ...))
)
)
}
Toutes ces modifications appliquées à nos données, avec le code ci-dessous, nous donne la Figure 4.
# Nous pouvons aussi ajouter un alignement pour chaque colonne avec un vecteur de 'char'
align_column=c("l","r","r","r","c","r","c","l","r","r","r","r","r","r","r","r")
widget_formattable = formattable(tab_reduce, align=align_column, list(
peptide_sequence = none_formatter(),
strand = formatter('span', style=style(font.weight = "bold")),
spliced = icon_formatter(),
best_IC50 = color_tile('lightblue', 'white'),
gene_id = none_formatter(),
gene_FPKM = color_bar_NA('red'),
mascot_score = color_tile('white', 'orange'),
area(col=c(intensity_1, intensity_2, intensity_3, intensity_4)) ~ normalize_bar("pink")
))
widget_formattable

Figure 4. Whaou!!!
Sauvegarder en fichiers HTML, JPEG, PNG ou PDF:
La librairie formattable permet d'exporter le tableau seulement dans un fichier au format HTML. Cependant, il existe une solution qui permet de convertir le tableau au format JPEG, PNG or PDF. Cette solution est un peu plus complexe et nous allons commencer par la version HTML.
Encore une fois, on nous simplifie la tâche. Il suffit de remplacer la fonction 'formattable' par 'format_table' pour obtenir le texte HTML de notre tableau. À ce texte, il faut ajouter un 'html_header' pour préciser le style css, c'est-à-dire le style visuel que nous voulons appliquer à notre tableau.
html_header="
<head>
<meta charset=\"utf-8\">
<meta name=\"viewport\" content=\"width=device-width, initial-scale=1\">
<link rel=\"stylesheet\" href=\"https://maxcdn.bootstrapcdn.com/bootstrap/3.3.7/css/bootstrap.min.css\">
</head>
<body>
"
html_table = format_table(tab_reduce, align=align_column, list(
peptide_sequence = none_formatter,
strand = formatter('span', style=style(font.weight = "bold")),
spliced = icon_formatter,
best_IC50 = color_tile('lightblue', 'white'),
gene_id = none_formatter,
gene_FPKM = color_bar_NA('red'),
mascot_score = color_tile('white', 'orange'),
area(col=c(intensity_1, intensity_2, intensity_3, intensity_4)) ~ normalize_bar("pink")
))
write(paste(html_header, html_table, sep=""), "./Supplementary_table_2_CrypticMAPSs.html")
Cela fonctionnne sans le 'html_header', mais dans ce cas, nous aurions perdu les icônes et le tableau aurait été affiché dans un format plus 'spartiate'.
Il ne nous reste plus qu'à finir, avec l'exportation au format JPEG, PNG et PDF. Pour ajouter cette fonctionnalité, nous avons besoin de la fonction 'export_table' et de deux librairies. Commençons par les installer:
install.packages('htmltools')
install.packages('webshot')
library(htmltools)
library(webshot)
webshot::install_phantomjs()
# Exporter le tableau en PNG, PDF ou JPEG
# Voir: https://github.com/renkun-ken/formattable/issues/26
export_formattable <- function(f, file, width = "100%", height = NULL,
background = "white", delay = 0.2)
{
w <- as.htmlwidget(f, width = width, height = height)
path <- html_print(w, background = background, viewer = NULL)
url <- paste0("file:///", gsub("\\\\", "/", normalizePath(path)))
webshot(url,
file = file,
selector = ".formattable_widget",
delay = delay)
}
Maintenant nous pouvons exporter le tableau dans le format voulu avec l'objet 'widget_formattable', comme ceci:
export_formattable(widget_formattable, "./Supplementary_table_2_CrypticMAPSs.png")
# Pour le PDF, les couleurs ne sont pas disponibles. C'est un problème connu.
# Voir: https://github.com/renkun-ken/formattable/issues/53
export_formattable(widget_formattable, "./Supplementary_table_2_CrypticMAPSs.pdf")
export_formattable(widget_formattable, "./Supplementary_table_2_CrypticMAPSs.jpeg")
Pour conclure:
Cette librairie est très efficace pour afficher un data.frame dans un format pratique et ce, dans notre navigateur web préféré (comparé View() et head()). De plus, les fonctions déjà fournies: color_tile(), color_bar(), area() and normalize_bar() devrait vous permettre d'analyser vos données en un coup d'oeil. Si ce n'est pas suffisant, on peut facilement personnaliser les éléments graphiques. C'est un peu plus compliqué à réaliser mais avec les exemples présents dans cet article, vous devriez arriver à vos fins. Pour les lecteurs de l'IRIC, il est aussi possible de demander l'aide de votre plate-forme favorite ;). À travers cet article, nous avons aussi identifié qu'il était impossible d'exporter en couleurs au format PDF. En attendant un correctif, il est possible d'exporter au format PNG et d'utiliser un autre logiciel pour transformer le fichier en PDF ou d'utiliser le format HTML.
J'espère que vous avez apprécié cet article et je vous souhaite beaucoup de plaisir avec formattable.


Laisser un commentaire