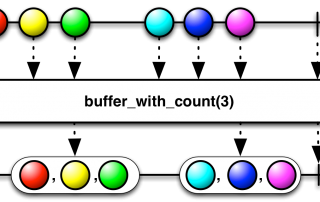
Documentez votre travail par l’ajout de paramètres à vos scripts shell
À un certain moment dans votre carrière de bioinformaticien, vous aurez à écrire des scripts shell, c'est inévitable ! Alors discutons d'une stratégie qui vous permettra à la fois de rendre vos scripts ré-utilisables et de garder une trace des paramètres utilisés pour générer une série de résultats. (Avertissement: les procédures décrites ci-bas ont été testées à l'aide du shell BASH, des modifications pourraient s'avérer nécessaires si vous utilisez un shell différent.) Il existe plusieurs techniques pour passer des paramètres [...]