L’application de méthodes statistiques forme une grande partie de la bio-informatique. En plus de méthodes classiques, certaines techniques d’apprentissage machine (notamment, des techniques de clustering telles k-means) sont aussi régulièrement appliquées sur des données cliniques et biologiques.
Quelques-unes de ces techniques telles les réseaux de neurones ont récemment connu un grand succès en reconnaissance d’images et traitement du langage naturel. Malheureusement, ces techniques performent mal sur de petits jeux de données ayant un grand nombre de dimensions, un type de jeux de données fréquemment rencontré à la plateforme.
L’introduction de données synthétiques créées artificiellement à partir des données originales permet de mitiger le débalancement entre la dimensionnalité du jeu de données et le nombre d’échantillons. Un plus grand nombre d’échantillons permet non seulement d’améliorer la performance, mais aussi la généralisation du modèle.
Création d’échantillons synthétiques de séquençage RNASeq
Un jeu de données d’un projet d’analyse d’expression de gènes issu de données RNASeq fut choisi comme candidat pour tester l’ajout de données synthétiques.
Pour ce jeu de données, la création d’un nouvel échantillon synthétique se fait par une simple méthode de bootstrap sur les reads d’un fichier d’alignement tophat en format BAM. Les reads sont ré-échantillonés avec remplacement à l’aide d’un script Python utilisant les outils de manipulation samtools par l’entremise de la librairie Pysam. Le fichier de sortie repasse ensuite par le pipeline d’analyse d’expression (cuffquant / cuffnorm) afin d’extraire le nouveau profil d’expression.
Voici un aperçu simplifié du script en question.
import pysam
import random
bam_in = "original.bam"
bam_out = "synthetic.bam"
bam = pysam.AlignmentFile(bam_in)
read_table = {}
for idx, read in enumerate(bam.fetch()):
read_table[read.qname] = 0
# Échantillonage
nb_reads = len(read_table)
read_list = read_table.keys()
for _ in range(0, nb_reads):
pick = read_list[random.randrange(nb_reads)]
read_table[pick] += 1
# Écriture du nouveau fichier basé sur l'échantillonage
new_bam = pysam.AlignmentFile(bam_out, "wbu", template=bam)
for idx, read in enumerate(bam.fetch()):
for x in range(0, read_table[read.qname]):
read.qname += "_%s" % x
new_bam.write(read)
new_bam.close()
Résultats
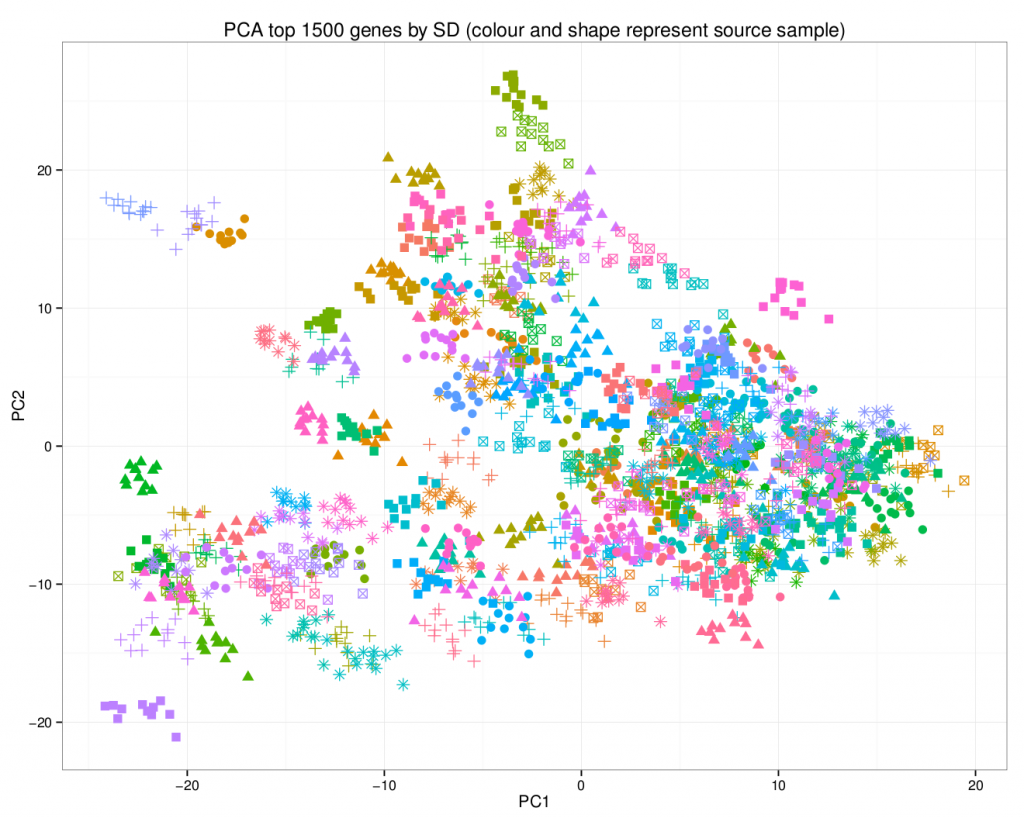
Une analyse en composantes principales du jeu de données permet de mieux visualiser l’ajout des données synthétiques.
Dans ce graphique, les points avec la même combinaison couleur/forme sont générés à partir du même échantillon. Notez à quel point ils se rassemblent.


Laisser un commentaire