Bonjour à tous,
aujourd’hui, nous allons voir une extension de la librairie ggplot2: cowplot
Some helpful extensions and modifications to the ‘ggplot2’ package. In particular, this package makes it easy to combine multiple ‘ggplot2’ plots into one and label them with letters, e.g. A, B, C, etc., as is often required for scientific publications.
Comme on peut le lire dans la description, cette librairie permet de créer des figures avec plusieurs graphiques (plots), mais pas uniquement. Il est aussi possible de l’utiliser pour créer des graphiques plus complexes et ce, en écrivant peu de lignes de code. Nous allons voir comment à travers un petit exemple (vous pouvez suivre l’exemple pas à pas ou regarder le code dans son entier ici)
Charger les librairies et créer quelques donnée
Pour cet exemple, nous avons besoin de charger les librairies: ggplot2 et cowplot ; et de générer des valeurs d’expression pour deux gènes dans deux groupes d’échantillons. Pour rendre cet exemple un peu plus intéressant, le premier gène aura une expression significativement différente entre les deux groupes, mais non le second.
library("ggplot2")
library("cowplot")
g1 = c(rnorm(200, mean=350, sd=100), rnorm(200, mean=700, sd=100))
g2 = c(rnorm(200, mean=350, sd=100), rnorm(200, mean=500, sd=100))
group = as.factor(rep(c(1,2), each=200))
df_exp = data.frame(G1=log2(g1 + 1) , G2=log2(g2 + 1), GROUP=group)
Créer chaque plot séparément
Comme d’habitude, nous commençons par afficher chaque information dans des graphiques distincts. L’astuce ici, c’est de sauver chaque graphique dans des variables afin de permettre de leur réutilisation ou leur modification dans les étapes suivantes. Pour cet exemple, nous avons décidé d’afficher l’expression des gènes sous la forme d’un nuage de points et d’afficher la distribution de chaque groupe et de chaque gène.
gg_scatter = ggplot(df_exp, aes(G1, G2, color=GROUP, shape=GROUP)) + geom_point(alpha=.8)
gg_dist_g1 = ggplot(df_exp, aes(G1, fill=group)) + geom_density(alpha=.5)
gg_dist_g1 = gg_dist_g1 + ylab("G1 density")
gg_dist_g2 = ggplot(df_exp, aes(G2, fill=group)) + geom_density(alpha=.5)
gg_dist_g2 = gg_dist_g2 + ylab("G2 density")
Différentes façons de regrouper vos graphiques, avec plot_grid
Maintenant, il faut regrouper nos 3 graphiques en 1 seul. La premère façon consiste à les concaténer en ajoutant des étiquettes pour les identifier dans un texte principal ou dans la légende. Avec cowplot, cela se fait très facilement en une ligne de code en utilisant la fonction plot_grid et en spécifiant quelques paramètres: nrow/ncol, labels:
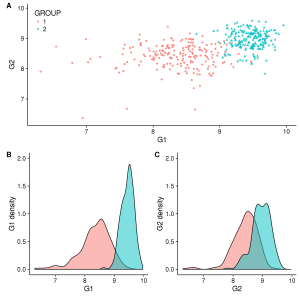
plot_grid(gg_scatter, gg_dist_g1, gg_dist_g2, nrow=1, labels=c('A', 'B', 'C')) #Or labels="AUTO"
Figure 1. A)Comparaison de 2 groupes en fonction de l’expression de 2 gènes. B)Distribution de G1. C)Distribution de G2.
Ici, on peut remarquer qu’il n’est pas nécessaire de dupliquer certaines informations (comme la legende) et que les graphiques seraient plus faciles à interpréter si la même echelle était appliquée sur tous les axes partagés par les graphiques. Ceci peut être rapidement corrigé, avec le code ci-dessous:
#Éviter d'afficher la légende plusieurs fois
gg_dist_g1 = gg_dist_g1 + theme(legend.position="none")
gg_dist_g2 = gg_dist_g2 + theme(legend.position="none")
#Homogenise scale of shared axes
min_exp = min(df_exp$G1, df_exp$G2) - 0.01
max_exp = max(df_exp$G1, df_exp$G2) + 0.01
gg_scatter = gg_scatter + ylim(min_exp, max_exp)
gg_scatter = gg_scatter + xlim(min_exp, max_exp)
gg_dist_g1 = gg_dist_g1 + xlim(min_exp, max_exp)
gg_dist_g2 = gg_dist_g2 + xlim(min_exp, max_exp)
gg_dist_g1 = gg_dist_g1 + ylim(0, 2)
gg_dist_g2 = gg_dist_g2 + ylim(0, 2)
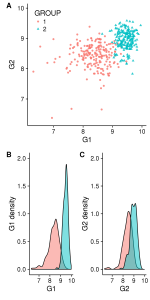
Il peut aussi être envisagé de déplacer B et C sur une autre ligne pour structurer notre figure en fonction du type de graphe. Comme toujours, il existe plusieurs façons de réaliser cette modification, ci-dessous nous allons créer chaque ligne séparément et les réunir en 1 colonne à l’aide de la fonction plot_grid (de cowplot) :
first_row = plot_grid(gg_scatter, labels = c('A'))
second_row = plot_grid(gg_dist_g1, gg_dist_g2, labels = c('B', 'C'), nrow = 1)
gg_all = plot_grid(first_row, second_row, labels=c('', ''), ncol=1)
#Afficher la légende
gg_scatter = gg_scatter + theme(legend.justification=c(0, 1), legend.position=c(0, 1))
 |
 |
Sauvegarder votre figure dans un fichier
Pour sauver votre figure dans un fichier et être certain que chaque graphique est redimensionné correctement en fonction de la taille globale de votre figure, cowplot propose d’utiliser la fonction save_plot, avec les paramètres base_height/base_width et ncol/nrow.
#Avec ncol=nrow=1, on spécifie que la figure (gg_all) est en un bloc avec un hauteur de base de 8 (base_height=8).
#Pour A, on doit redimensionner pour s'ajuster à la largeur de B+C (calculée en fonction de la hauteur)
save_plot("./all_v2.png", gg_all, base_height=8, ncol=1, nrow=1) #Figure 2
#Ici, votre figure a 2 blocs séparés par rangée et chaque rangée a une hauteur de base de 4 (base_heigth=4)
#B et C doivent être redimensionnés pour s'ajuster à la largeur de A.
save_plot("./all_v3.pdf", gg_all, base_height=4, ncol=1, nrow=2) #Figure 3
Vous avez pu noter que l’extension dans le nom du fichier suffit à spécifier le type de fichier de sortie désiré. Il n’est plus nécessaire d’adapter notre code en fonction du type de fichier souhaité (png(), pdf(), …).
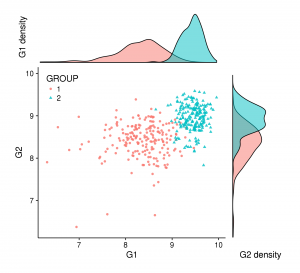
Tout en 1!
Parfois, il est possible d’agréger tous les graphiques en un seul plus attrayant et parlant, comme la figure en en-tête de cet article. Mais (pour certains d’entre nous) ce genre de graphique peut être complexe à réaliser avec des logiciels comme Illustrator (et autres). Si c’est votre cas, ggplot2+cowplot est une alternative à envisager sérieusement. En effet, cette figure peut être réalisée avec quelques lignes supplémentaires au code ci-dessus:
#Nous devons interchanger les axis de gg_dist_g2
gg_dist_g2 = gg_dist_g2 + coord_flip()
#Retrait des axes dupliqués
gg_dist_g1 = gg_dist_g1 + theme(axis.title.x=element_blank(),
axis.text=element_blank(),
axis.line=element_blank(),
axis.ticks=element_blank())
gg_dist_g2 = gg_dist_g2 + theme(axis.title.y=element_blank(),
axis.text=element_blank(),
axis.line=element_blank(),
axis.ticks=element_blank())
#Modification des marges, c(top, right, bottom, left), pour réduire la distance entre les graphiques
#et alignement du graphe de densité de G1 au nuage de points.
gg_dist_g1 = gg_dist_g1 + theme(plot.margin = unit(c(0.5, 0, 0, 0.7), "cm"))
gg_scatter = gg_scatter + theme(plot.margin = unit(c(0, 0, 0.5, 0.5), "cm"))
gg_dist_g2 = gg_dist_g2 + theme(plot.margin = unit(c(0, 0.5, 0.5, 0), "cm"))
#Ici, combinaison de tous les graphiques et ajustement de la taille des graphes de densité
#en utilisant rel_heights
first_col = plot_grid(gg_dist_g1, gg_scatter, ncol = 1, rel_heights = c(1, 3))
second_col = plot_grid(NULL, gg_dist_g2, ncol = 1, rel_heights = c(1, 3))
perfect = plot_grid(first_col, second_col, ncol = 2, rel_widths = c(3, 1))
save_plot("./perfect.png", perfect, base_height=6)
Pour conclure
Cet article n’est qu’une introduction à la librairie cowplot. Nous n’avons mentionné que 2 fonctions! Dans cette librairie vous pouvez notamment utiliser draw_plot qui permet d’afficher un graphique à une coordonnée précise. Je l’utilise quand j’ai besoin de superposer plusieurs graphiques. Ça peut notamment servir à changer la couleur de l’arrière plan en fonction d’un paramètre. Ce n’est qu’un autre exemple parmi de nombreux autres, ainsi si vous êtes curieux d’en savoir plus je vous recommande de lire les vignettes et le manuel ici et de laisser parler votre imagination.


Laisser un commentaire