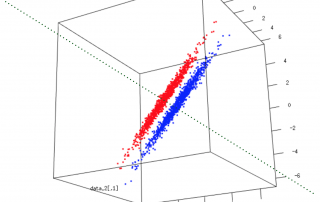
Tutoriels de réduction de dimensionalité: 1- Analyse de composantes principales
Comprendre la réduction de dimensionalité Si vous utilisez de larges jeux de données (transcriptomes, séquençage de génome, protéomes), tôt ou tard, vous tomberez sur quelque chose qui porte le nom "d'analyse de composantes principales" (Principal Components Analysis, en anglais, abrévié PCA). PCA est une méthode de réduction de dimensionalité, une famille large de méthodes qui font exactement ce que leur nom dit: elles réduisent la dimensionalité. Mais qu'est-ce que ça veut dire? Qu'est-ce qu'une dimension et pourquoi on voudrait les [...]