Mise à jour 27/08/2018
J’ai déjà introduit la librairie Python pandas en comparant certaines de ses fonctions à leurs fonctions équivalentes en R. Pandas est une librairie qui rend Python beaucoup plus facile à utiliser pour faire de l’exploration et de la visualisation de données (presque autant que R). La librairie permet de manipuler des dataframes très facilement (elle est construite par-dessus numpy). Elle a beaucoup évolué ces dernières années et la communauté d’utilisateurs a aussi beaucoup grossie. R offre certainement plus de modules statistiques spécialisés, mais pandas est intéressant car il est maintenant intégré dans un bon nombre de packages, notamment dans le package de chémoinformatique, rdkit. Utiliser pandas pour analyser des données en Python rend l’expérience plus agréable, comparativement à utiliser Python seul.
R et Python sont deux langages intéressants, capables d’effectuer les mêmes tâches. Dans les deux cas, plusieurs libraries sont disponibles et viennent ajouter des « fonctionnalités ». Par exemple, charger les données de deux fichiers textes contenant 176241 rangées et, respectivement, 151 et 574 colonnes prend significativement moins de temps avec Python : environ 1 à 2 minutes versus 19 minutes en R en utilisant la fonction de base
read.delim
. Toutefois, il existe des librairies qui permettent de charger des données en R beaucoup plus rapidement, en 30 secondes ou moins pour ce cas spécifique. Les deux langages sont donc des options appropriées pour manipuler de larges jeux de données.
Ci-dessous, je revisite l’exercice de « traduction » entre R et Python/pandas.
Extraits d’un exemple réel
Mon but est de comparer directement les profils d’expression de gènes de patient provenant de TCGA et de Leucegene; tous les résultats de séquençage ayant été traités de la même façon. Les niveaux d’expression sont des tpm dérivés du programme kallisto pour le fichier d’annotation GRCh38.84. Les fichiers d’entrées contiennent les expressions des transcrits des gènes qui sont identifiés par leur Ensembl ID. J’ai fait la comparison entre les deux ensembles de données en R et Python/pandas (je testais le kernel R de Jupyter Notebook). Par souci de simplicité, j’ai sélectionné de petites sections de code parmi l’analyse complète.
1. Lecture des fichiers d’entrées
R :
tcga <- read.delim('TCGA_LAML_kallisto.txt', header=TRUE)
leuc <- read.delim('Leucegene_GRCh38.84.txt', header=TRUE) Python :
tcga = pandas.read_csv('TCGA_LAML_kallisto.txt', sep='\t', index_col=0,low_memory=False)
leuc = pandas.read_csv('Leucegene_GRCh38.84.txt', sep='\t', index_col=0,low_memory=False) 2. Définition d’une fonction
La fonction head2 retourne les n première(s) rangée(s) et n première(s) colonne(s).
R :
# En R, la première position est 1.
head2 <- function(mx, n=3) { return (mx[1:n, 1:n]) }Python :
# En Python, la première position est 0.
def head2 (mx, n=3) :
return mx.iloc[0:n,0:n]3. Vérifier la taille du jeu de données
R :
print(dim(tcga))
print(dim(leuc))Python :
print tcga.shape
print leuc.shape4. Reformatage du nom des colonnes
Les noms des colonnes ont été générés par le pipeline et contiennent le chemin vers le répertoire où se trouvaient les différents fichiers traités. Pour le jeu de données de Leucégène, les noms de colonnes ressemblent à
/data/DSP001/02H053/transcriptome/kallisto/GRCh38.84/abundance.tsv. Pas très convivial. Pour modifier ces noms de colonnes, nous utilisons des méthodes spécifiques aux chaînes de caractères.
R :
# La fonction colnames() est utilisée pour récupérer les noms des colonnes d'une matrice ou d'un dataframe.
# En R, les barres sont remplacées par un '.' et un 'X' est ajouté au début des noms
# de colonnes qui ne commencent pas par une lettre.
# Nous remplaçons d'abord les parties communes à tous les noms.
colnames(leuc) <- gsub('X.data.', '', colnames(leuc))
colnames(leuc) <- gsub('transcriptome.kallisto.GRCh38.84.abundance.tsv', '', colnames(leuc))
# La partie DSP001/02H053 change entre les échantillons. Nous voulons récupérer le
# id de l'échantillon, dans ce cas-ci, 02H053 (DSP001 étant le id du projet)
# Pour cela, nous séparons la chaîne de caractères et retournons le deuxième élément,
# qui deviendra le nom de la colonne.
colnames(leuc) <- apply(as.matrix(colnames(leuc)), c(1,2), function(x) {return(strsplit(x, split='\\.')[[1]][2])})Python :
# .columns est utilisé pour obtenir les noms des colonnes d'un dataframe.
# En Python, nous pouvons utiliser ce qu'on appelle une list comprehension au lieu
# d'utiliser une boucle for pour itérer parmi les éléments d'une liste.
# Par exemple, [x+1 for [1,2,3,4]] ajoutera 1 à tous les élements de la liste donnée.
# Ici, nous remplaçons la partie commune des noms et séparons la chaîne de caractères
# restante pour retourner le deuxième élément.
leuc.columns = [x.replace('/data/', '').replace('/transcriptome/kallisto/GRCh38.84/abundance.tsv', '') \
.split('/')[1] for x in leuc.columns]5. Générer un graphique
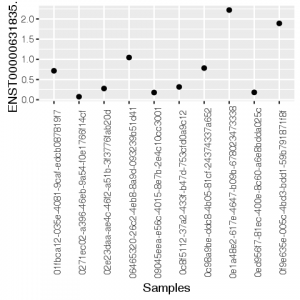
library(ggplot2)
mx <- as.data.frame(t(tcga[105,1:10]))
mx[,1] <- as.numeric(as.vector(mx[,1]))
mx$Samples <- as.factor(rownames(mx))
ggplot(mx, aes(x=Samples, y=`ENST00000631835.1`))+geom_point()+theme(axis.text.x = element_text(angle = 90))
Python :
## Il existe un module ggplot2 pour Python, mais ici
## nous utilisons les fonctions de matplotlib qui sont intégrées dans pandas
## Nous utilisons le style graphique de ggplot2 par contre.
import matplotlib
matplotlib.style.use('ggplot')
tcga.ix[["ENST00000631835.1"], 0:10].astype('float').T.plot(marker='o', rot=90)
Voici ce que ça donne :
| R/ggplot2 | Python/Pandas |
|---|---|
 |
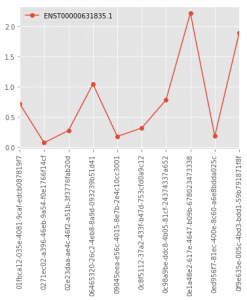 |
Choisir un langage de programmation plutôt qu’un autre dépend de la tâche à accomplir, mais est aussi lié à un choix personnel. Toutefois, en maîtrisant plus d’un langage, on se donne nécessairement plus de flexibilité.


Laisser un commentaire